 ,
Isabella Betancourth-Arteaga1
,
Isabella Betancourth-Arteaga1  ,
Adriana Lucía Romero-Martínez1
,
Adriana Lucía Romero-Martínez1  ,
Mónica Chávez-Vivas2
,
Mónica Chávez-Vivas2 
Introducción. La problemática alrededor de la resistencia a los antibióticos se intensifica por la presencia de patógenos resistentes en alimentos de origen animal. Objetivo. Presentar el estado de la prevalencia de bacterias resistentes a antibióticos (BRA) y los principales genes de resistencia a antibióticos (GRAs) que se reportan en alimentos de origen animal y en animales destinados al consumo humano. Materiales y métodos. Se realizó una revisión sistemática basada en la guía PRISMA, empleando las bases de datos: Science Direct, Redalyc, Scopus, Hinari, Scielo, Dialnet, PLOS, ProQuest, Taylor, Lilacs y PubMed/ Medline con estudios originales realizados entre enero de 2017 y abril 2023. Resultados. Un total de 2620 estudios fueron identificados y 71 estudios cumplieron los criterios de inclusión. La carne de res, leche cruda/productos lácteos no pasteurizados y las heces de animales de granja fueron las muestras más estudiadas. Las BRAs más frecuentes fueron Escherichia coli productora de β-lactamasas de espectro extendido (BLEE), Salmonella spp. resistente a múltiples fármacos (MDR) y Stahylococcus aureus resistente a meticilina (SARM). Los GRAs más reportados fueron bla, tet y sul mediados por plásmidos e integrones, principalmente. Conclusiones. En esta revisión sistemática se encontró, que los aislamientos de E. coli, Salmonella spp. y S. aureus son los que más frecuentemente presentaron resistencia a la tetraciclina ampicilina y el sulfametoxazol/ trimetoprima con el predominio de los genes bla, tet y sul, que están siendo diseminados por elementos genéticos móviles entre bacterias y a humanos a través de clones zoonóticos con una alta estabilidad en el tiempo. Arch Latinoam Nutr 2023; 73(4): 313-327.
Palabras clave: resistencia, antibióticos, alimentos, ganado, aves de corral.
Introduction. The problem around antibiotic resistance is intensified by the presence of resistant pathogens in foods of animal origin. Objective. Present the state of the prevalence of antibiotic resistant bacteria (ARB) and the main antibiotic resistance genes (AGRs) that are reported in foods of animal origin and in animals intended for human consumption. Materials and methods. A systematic review was carried out based on the PRISMA guide, from the Science Direct, Redalyc, Scopus, Hinari, Scielo, Dialnet, PLOS, ProQuest, Taylor, Lilacs and PubMed/Medline databases with original studies carried out between January 2017 and April of 2023. Results. A total of 2620 studies were identified, and 71 studies met the inclusion criteria. Beef, raw milk/unpasteurized dairy products, and farm animal feces were the most studied samples. The most common resistant bacteria were extended-spectrum β-lactamase (ESBL)-producing Escherichia coli, Salmonella spp. multidrug resistant (MDR) and methicillin resistant Stahylococcus aureus (MRSA). The AGRs most reported were bla, tet and sul, mediated mainly through plasmids and integrons. Conclusions. In this systematic review it was found that the isolates of E. coli, Salmonella spp. and S. aureus are the ones that most frequently presented resistance to tetracycline ampicillin and sulfamethoxazole/ trimethoprim with a predominance of the bla, tet and sul genes, which are being disseminated by mobile genetic elements between bacteria and humans through zoonotic clones with high stability over time. Arch Latinoam Nutr 2023; 73(4): 313-327.
Keywords: resistance, antibiotics, foods, livestock, poultry.
https://doi.org/10.37527/2023.73.4.006
Autor para la correspondencia: Mónica Chávez Vivas. e-mail: [email protected], [email protected]
La sociedad se encuentra en una lucha constante contra la resistencia a los antibióticos, debido a la pérdida de efectividad de los antibióticos (1). La problemática se intensifica por la presencia de patógenos resistentes fuera del entorno clínico, especialmente detectados en animales de consumo humano (2,3).
Al utilizar de manera habitual los antibióticos en los criaderos de animales destinados a la alimentación para tratar y prevenir enfermedades infecciosas o para promover su crecimiento, se favorece la diseminación de bacterias resistentes a los antibióticos (BRA) y genes de resistencia a los antibióticos (GRA) entre los animales, sus productos y el medio ambiente terrestre y acuático (4). Las muestras fecales de animales de granja pueden contener BRA y GRA facilitando su diseminación en el suelo, los cultivos destinados a la alimentación y en los sistemas de aguas (4-11).
En este sentido, con las actividades agrícolas, el sacrificio animal, la producción de alimentos de origen animal y el transporte de animales destinados a la alimentación se puede introducir BRA y GRA en la cadena alimentaria (11-19).
Algunos investigadores sugieren que antibióticos residuales en la carne cruda puede también contribuir aldesarrollo de BRA en la microbiota intestinal humana (14, 15,18). Esto se debe a que los humanos y el ganado comparten bacterias patógenas y comensales y porque se usan las mismas clases de antibióticos en medicina humana y veterinaria. La resistencia a los antibióticos también pone en peligro la eficacia de los antibióticos para curar a los animales de infecciones bacterianas que amenazan su salud, bienestar y productividad (20).
El objetivo de esta revisión sistemática es describir el estado de resistencia a los antibióticos de bacterias aisladas de alimentos de origen animal y de animales de consumo humano y la distribución de la resistencia de los genes de resistencia a lo largos del mundo. En segundo lugar, brindamos una evaluación de la transmisión de bacterias resistentes de animales a humanos.
Esta revisión se llevó a cabo siguiendo la pauta del informe PRISMA (Preferred Reporting Items for Systematic Reviews and Meta- analysis). La información se recopiló mediante la búsqueda de artículos publicados en bases de datos electrónicas y servicios de indexación como PubMed/ Medline, ScienceDirect (Elsevier), Scopus, Scielo, PLOS, Hinari, Redalyc, Dialnet, Taylor, ProQuest y Lilacs. Las combinaciones de términos de búsqueda utilizados fueron "bacterias resistentes a los antibióticos”, “Genes resistentes a los antibióticos”, “alimentos de origen animal", “animales destinados a la producción de alimentos”, “animales de consumo humano”, “animales de alimentación”, "animales de granja''. Otros descriptores específicos de animales destinados a la alimentación fueron: “aves de corral”, “pollos”, "cerdos", "porcinos”, “ganado”, “ganado de carne”, “ganado lechero”, “pescado”. Se aplicaron los conectores lógicos booleanos (AND/Y, OR/O) para la adecuada búsqueda e identificación de registros de acuerdo con la pregunta de investigación.
Los artículos incluidos cumplieron los siguientes criterios: 1) la estrategia de búsqueda estuvo restringida al idioma inglés y español; 2) debía ser un estudio de investigación original o un meta-análisis publicado en una revista científica; 3) información que describa la prevalencia de BRAs y GRAs aislados de animales de consumo humano o alimentos de origen animal;
4) estudios publicados entre enero de 2017 y abril de 2023. Se excluyó los artículos de revisión, las revisiones sistemáticas, las editoriales y las declaraciones de política.
Un total de 2620 registros se encontraron en las bases de datos analizadas; sin embargo, 71 artículos cumplieron los criterios de inclusión propuestos (6-76) (Figura 1).
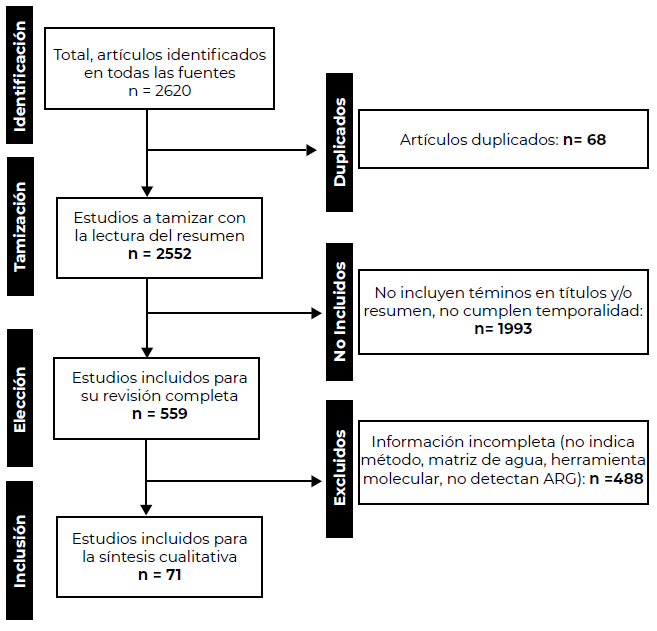
El mayor número de publicaciones se recopilaron de Asia (20 artículos), especialmente en China con ocho estudios, seguido de África con 16 estudios publicados.
Los reportes de BRAs y GRAs que se reportaron con mayor frecuencia provenían de muestras de carne de res (24 estudios), leche cruda/productos lácteos no pasteurizados (23 estudios) y heces de animales de granja (17 estudios).
Si bien los métodos genéticos y genómicos para identificar y monitorear la resistencia a los antimicrobianos están experimentando un rápido desarrollo, aún los métodos fenotípicos basados en la difusión en disco siguen siendo los más usados cómo se observa en la Tabla 1.
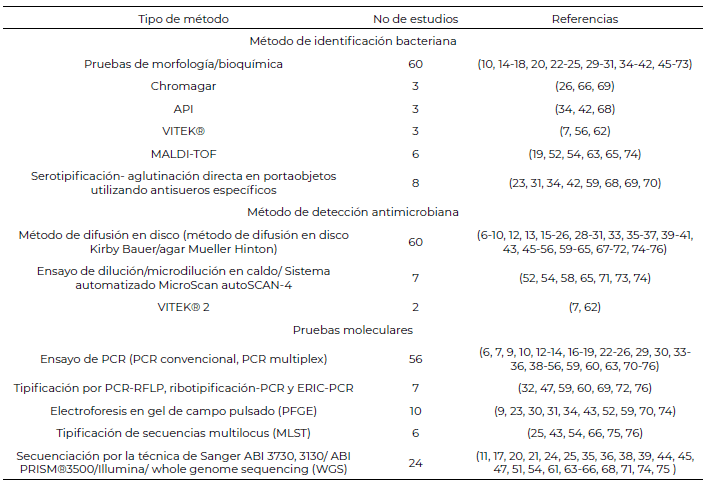
Los datos de resistencia a los antimicrobianos se tomaron directamente de cada estudio y la interpretación de las mediciones de las concentraciones inhibitorias o zonas de inhibición en términos de resistencia, resistencia intermedia y susceptible estuvo sujeta a la interpretación del autor, siguiendo en la mayoría de los casos los criterios establecidos por el Instituto de Estándares Clínicos de Laboratorio (CLSI) (en 47 estudios) o por el Comité Europeo de Pruebas de Susceptibilidad Antimicrobiana (EUCAST) (en 14 estudios).
El análisis de susceptibilidad antimicrobiana de los aislamientos bacterianos obtenidos a partir de muestras de hisopados rectales o cloacales, leche cruda/productos lácteos no pasteurizados y biopsias gástricas de animales de granja se basaron en función de los puntos de corte clínicos (21-36).
La mayoría de los estudios reportaron la resistencia a los antibióticos basados en criterios de la clínica humana, sólo tres publicaciones reportaron los patrones de susceptibilidad con puntos de corte específicos para medicina veterinaria como fue el caso de la evaluación de la susceptibilidad al ceftiofur que se realizó en aislamientos de Escherichia coli y Staphylococcus aureus obtenidos de leche cruda de vacas con mastitis bovina (38-40).
En términos generales, entre los estudios analizados, los reportes de bacterias resistentes a los antibióticos más frecuentes se realizaron para las bacterias Gram negativas, E. coli y Salmonella spp en 27 y 20 estudios, respectivamente y para bacterias Gram positivas, S. aureus, reportada en 14 estudios, especialmente en países de África, Asia y Europa (Figura 2A).
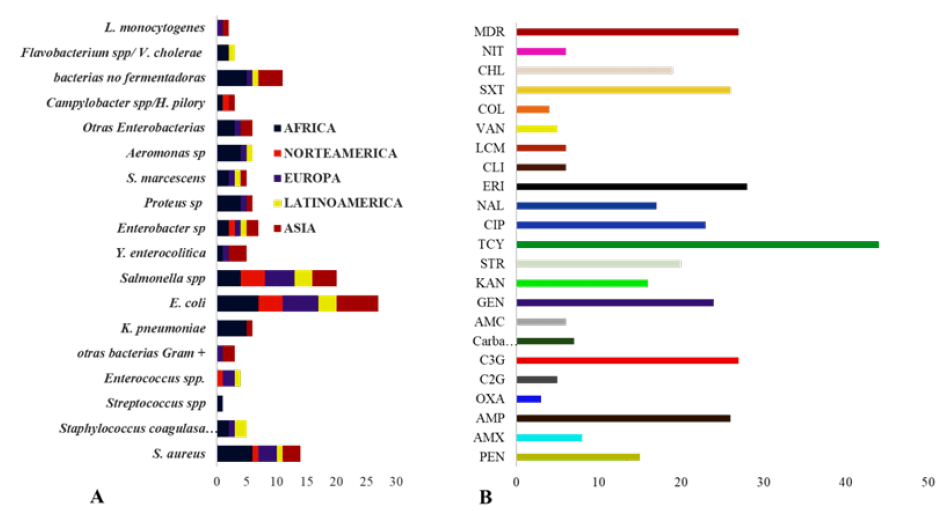
En estos casos, la tetraciclina fue el antibiótico con más reportes de resistencia (44 estudios), seguido de ampicilina y sulfametoxazol/ trimetoprima (26 estudios c/u) (Figura 2B).
Un aspecto relevante lo constituyó la presencia de bacterias MDR (con resistencia simultánea a más de tres clases de antibióticos) que fueron reportadas en 38 artículos (Figura 2B). El mayor número de aislamientos MDR se reportó en heces, hisopados cloacales de pollos e hisopados rectales de ganado y cerdos (9, 24, 30, 35, 47-50, 67, 71, 76). En los alimentos de origen animal, los aislamientos MDR se reportaron principalmente en carne cruda y productos cárnicos (12, 40, 41, 44, 46, 55, 62, 69) y en leche cruda/productos lácteos no pasteurizados (12, 30, 41, 49, 62,69).
E. coli y Salmonella spp. fueron los aislamientos MDR más reportados en muestras cloacales y rectales de animales de granja especialmente en países europeos (18, 53, 56, 70), africanos (22, 42, 45, 48), asiáticos (15, 61) y en los Estados Unidos (23,24).
En muestras de carne de pollo en mercados de Bangladesh, los aislamientos de E. coli-MDR fueron del 50% (14). Del mismo modo, Jaja et al (46) reportaron una alta prevalencia de aislamientos de E. coli-MDR en expendios de carnes en Sudáfrica.
Otra de las enterobacterias reportadas con frecuencia como MDR fue Yersinia enterocolitica, la cual se detectó en el 90% de los hisopados rectales de cerdos y productos porcinos en Malasia (34), estos animales han sido implicados como un importante reservorio de la bacteria en la cavidad oral, que luego se excreta en las heces (31, 34, 54). También se reportó un 70% de estos aislamientos en muestras de carne que se encontraban en sitios de venta al por menor en Egipto (44) y en el 63% de las muestras de mariscos tomadas en tres mercados centrales de Tailandia (68).
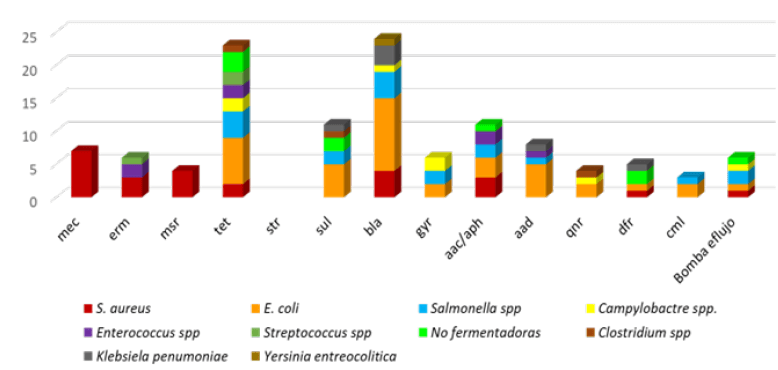
Un total de 50 publicaciones reportaron GRAs en alimentos crudos de origen animal o en animales destinado al consumo humano, la mayoría de ellas en Asia (45 estudios), seguido de África (25 estudios) (Figura 3). Las publicaciones hacen referencia principalmente a los genes bla (codifica para la β-lactamasa y confiere resistencia a antibióticos β-lactámicos) en 24 estudios, genes tet (confiere resistencia a tetraciclinas) en 23 estudios, genes sul (confiere resistencia a sulfamidas) en 11 estudios, genes aac(6')-aph(2") (confiere resistencia a aminoglucósidos) y genes add (coçdifica por la enzima 3''(9)-O-aminoglucósido adenililtransferasa, confiere resistencia aminoglucósidos) en 8 estudios. Los genes bla, tet y sul se reportaron principalmente entre los aislamientos de E. coli presentes en alimentos crudos de origen animal (14, 30, 44, 46, 58, 64, 65, 68) y en heces de animales de granja (8, 10, 11, 48-50).
Aunque se viene restringiendo el uso de antibióticos en animales productores de alimentos, especialmente cuando se emplea como agentes profilácticos o promotores del crecimiento (2-4), se continúa reportando bacterias patógenas con resistencia a los antibióticos aisladas de alimentos de origen animal especialmente E. coli, Salmonella spp y S. aureus. Sin embargo, el análisis de susceptibilidad a los antibióticos en la mayoría de los estudios analizados se basó en función de los puntos de corte clínicos. Silva et al (37), plantean que estos tipos de análisis podrían estar sesgados por las diferencias que existen entre animales y humanos, desde el punto de vista fisiológico, farmacocinético y farmacodinámico, por lo que se obtendrían datos imprecisos, incluso a clases de antimicrobianos de importancia crítica, como las cefalosporinas de cuarta generación y los carbapenémicos. Como alternativa a los puntos de corte clínicos, se propone establecer puntos de corte epidemiológicos (epidemiological cut-offs ECOFFS), en los que se utiliza la interpretación de resistencia normalizada (NRI, por sus siglas en inglés) de concentraciones inhibitorias mínimas o diámetros de zona de inhibición (IZD, por sus siglas en inglés) para identificar umbrales y distinguir las poblaciones bacterianas de tipo salvaje (WT por sus siglas en inglés) de las de tipo no salvaje (no WT por sus siglas en inglés) (5), pero los ECOFFS requieren ser estandarizado para cada especie animal.
Se destacan los reportes de. E. coli productora de β-lactamasas de espectro extendido (BLEE) (38, 50, 55, 58, 65, 74) y de S. aureus resistente a meticilina (SARM)(12, 25, 30, 40, 62) que son dos géneros motivo de preocupación en infecciones asociadas al entorno hospitalario (1, 2). La contaminación de los alimentos de origen animal con estas bacterias patógenas representa una gran amenaza para la salud pública, debido a que se plantea que los determinantes de resistencia a los antibióticos que transportan estas bacterias se puedan transferir al microbiota intestinal humana, además del riesgo de contraer enfermedades transmitidas por alimentos (46, 62, 73).
Las deposiciones (heces) de los animales de granja destinados a consumo humano fueron las fuentes principales de E. coli productores de BLEE (reportada en 10 estudios) y Salmonella spp. resistente a los antibióticos (reportada en 6 estudios), lo que constituyen motivo de preocupación porque sería una fuente potencial de patógenos resistentes y de GRA para los productos agrícolas y el suelo.
En países de la Unión Europea se destacó la presencia de aislamientos de Salmonella, principalmente en aves de corral, las especies más reportadas fueron S. Enteritidis, S Typhimurium y S. Kentucky (18, 53, 56, 70) con resistencia más frecuente a las tetraciclinas y a las quinolonas, y en los Estados Unidos con un 22% de resistencia a las fluoroquinolonas y entre 41-46% a las tetraciclinas (23, 24) coincidiendo con los reportes de la Unión Europea; aunque los aislamientos provenían también de ganado y cerdos. Los estudios revisados en África muestran que la problemática de resistencia de Salmonella está relaciona con cepas productores de BLEEs que se obtienen de muestras cloacales y rectales de animales de granjas comerciales de Nigeria (22), Egipto (42), Etiopía (45) y Sudáfrica (48).
En Asia, los reportes señalan una mayor prevalencia de aislamientos de Salmonella resistentes a varias clases de antibióticos como es el estudio realizado en productos cárnicos que se venden al por menor, abarcando la mayoría de las capitales provinciales de China en el que se determinó que los aislamientos de S. Weltevreden y S. Enteritidis presentaronaltastasasde resistencia a la tetraciclina (65,6%), ampicilina (45,4%), trimetoprim-sulfametoxazol (40,8%), estreptomicina (40,4%) y ácido nalidíxico (35,8%), mientras que, los aislamientos de S. Indiana mostraron resistencia a las cefalosporinas de espectro extendido (incluidas la ceftriaxona y la cefepima), ciprofloxacina y muchas otras clases de antibióticos (15). Resultados similares se reportan en los aislamientos obtenidos de muestras de leche cruda recolectadas en cinco distritos de la parte sur de la provincia de Punjab, sólo acloranfenicol y ofloxacina se registró mayor sensibilidad (61).
En las cepas de S. aureus, se encontró que el mayor número de reportes de SARM se efectuó a partir de muestras de carne (siete estudios) y de pescado (cuatro estudios) que se encontraban en los sitios de expendio, especialmente en países de África (12, 40, 41, 62). Sin embargo, también existe alta prevalencia de SARM en alimentos de origen animal en Europa (25, 37, 39), Estados Unidos (72) y en Asia (26, 64), lo que representa un enorme problema de salud pública en estas regiones.
Los Enterococcus son otras de las bacterias Gram positivas que se reportan en muestras de animales destinados al consumo humano y en sus productos, especialmente Enterococcus faecalis y E. faecium (73, 19, 20,36). Los Enterococcus son habitantes del intestino de animales destinados al consumo humano y pueden contaminar fácilmente los productos alimenticios si no se manejan adecuadamente. Algunos estudios señalan una alta prevalencia de Enterococcus resistentes a los antibióticos en alimentos fermentados, como es el caso de los reportes en muestras de quesos que se venden al por menor en tiendas de Eslovaquia y Hungría (fenotipo de resistencia: rifampicina 100%, vancomicina 85,7%, teicoplanina 71,4%, eritromicina 71,4%, minociclina 57,1%) (19). Así como en muestras de queso que se vende en bazares y supermercados locales en Ankara (Turquía) (fenotipo de resistencia: ácido nalidíxico 100%, kanamicina 98,6%, rifampicina 78,4%, ampicilina 48,8% y ciprofloxacina 45,5%) (20). Un análisis realizado en muestras de carne de cerdo fermentado que se ofrecen en los mercados de la provincia de Chiang Rai, Tailandia determinó los fenotipos con resistencia principalmente a ciprofloxacina (97,5%), eritromicina (78,2%) y tetraciclina (67,2%) (36).
En alimentos cárnicos procesados también se reporta cepas de Enterococcus resistentes. El estudio que incluyó muestras de carne de pollo, pavo molido, carne molida y chuletas de cerdo en ventas al por menor en varias ciudades de los Estados Unidos determinó aislamientos resistentes a penicilina, ciprofloxacina, gentamicina, kanamicina, tetraciclina y tylosina (73).
Todos estos antibióticos a los cuales se reportan habitualmente resistencia se han empleado en medicina veterinaria durante décadas como promotores de crecimiento o para prevenir o tratar infecciones (3, 4), lo que contribuye a fortalecer el aumento de bacterias resistentes en los productos alimenticios generados a partir de estos animales. En este sentido, el estudio realizado con muestras de hisopados rectales de cerdos en Argentina, demostró que la presencia de aislamientos de Enterococcus resistentes a la tetraciclina estaba relacionada con el uso generalizado de zinc y tetraciclina como promotores de crecimiento en la alimentación de estos animales. Esta práctica ejerce una presión selectiva en especies bacterianas zoonóticas y contribuye a la propagación de la resistencia a los antibióticos como lo sugieren los autores (57).
Por otra parte, algunos estudios no encontraron enterococos resistentes a algunos antibióticos en las muestras de alimentos analizadas, como es el caso de la vancomicina (20, 36, 73). Los autores atribuyen este resultado a las restricciones impuestas al empleo de la avoparcina (análogo de la vancomicina) como estimulador del crecimiento en países europeos y en Tailandia (20, 36) y al uso de la vancomicina en medicina veterinaria en los Estados Unidos (73).
Hay también una preocupación con respecto al reporte de aislamientos de Campylobacter resistentes a los antibióticos que probablemente vienen siendo seleccionados por el uso de antimicrobianos en la cría intensiva de animales, especialmente en la avicultura. Particularmente la resistencia a las fluoroquinolonas en los aislamientos obtenidos de las aves de corral (7, 47, 51, 66).
En esta revisión se encontró que la tetraciclina, ampicilina y sulfametoxazol/trimetoprima fueron los antibióticos en los que más frecuentemente se reportaron bacterias resistentes. Estos antimicrobianos se usan comúnmente en el ganado, cerdos y las aves de corral por la actividad que presentan contra una gran variedad de microorganismos patógenos, además son empleados para acelerar el aumento de peso y para prevenir o tratar brotes de enfermedades infecciosas (4). Campylobacter es considerado una bacteria que habitualmente coloniza el tracto intestinal en los animales destinados al consumo humano con gran capacidad de adquirir resistencia a los antibióticos. Coulidiaty et al (7) reportaron en granjas de pollos de Burkina Faso a Campylobacter resistente a ciprofloxacina y ácido nalidíxico (37,5%), ampicilina (50%) y tetraciclina (87,5%), que son los antibióticos más utilizados por los agricultores. Sithole et al (47) reportaron alta prevalencia de aislamientos de Campylobacter spp resistentes a la tetraciclina y ampicilina, que son antibióticos utilizados intensivamente en la producción porcina en Sudáfrica. Además, tiene la capacidad de transmitir sus determinantes de resistencia a los antibióticos a otras bacterias. En este sentido, Hull et al (51) comprobaron la transferencia de GRAs entre especies en C. coli y C. jejuni que habían sido aisladas de hisopados cloacales y rectales de animales de granja en Carolina del Norte y de carne cruda lista para la venta al por menor. Los autores plantean que este intercambio genético puede ayudar a la adaptación de estos patógenos, conducir a una mayor resistencia y aumentar la persistencia de Campylobacter en el procesamiento de alimentos. Varios investigadores hacen énfasis en la necesidad de hacer seguimiento a estas cepas resistentes por el riesgo que representan para la seguridad alimentaria, al diseminarse con facilidad tienen la potencialidad de causar brotes de enfermedades transmitidas por los alimentos y el agua (7, 47, 51, 55, 66).
En concordancia con estos resultados, entre los aislamientos de E. coli resistentes a los antibióticos obtenidos de carne de aves de corral, pollos de engorde y gallinas ponedoras en Bangladesh, el patrón de resistencia coincidió con los antibióticos de uso común, como la tetraciclina y otros (14). Un estudio realizado en granjas en Filipinas reportó alta similitud entre los aislamientos de E. coli obtenidos de muestras de carne de res, pollo, cerdo, huevos y leche cruda con aislamientos ambientales recolectadas de hisopos cloacales y rectales de animales. También se reportó la presencia de residuos de antibióticos entre restos alimenticios, por lo que los autores sugieren la posibilidad de ingresar al humano cepas resistentes y residuos de antibióticos mediante el consumo de estos tipos de alimentos (33).
En este sentido, en el estudio realizado en muestras de leche recolectadas de hatos lecheros ubicados en Karnataka, India, se detectó residuos de azitromicina y tetraciclina. La estabilidad de estos antibióticos sólo se logró reducir aplicando temperaturas entre 70oC y 100°C durante 24 horas o sometiéndolas a pH ácidos de 4–5, por lo que los autores sugieren que la detección cualitativa y cuantitativa de antibióticos en la leche debe realizarse estrictamente para garantizar su consumo seguro (16). Sin embargo, Brown et al. (13) realizaron análisis de leches no pasteurizada en lecherías de Kenia, y a pesar de la detección de residuos de los antibióticos β-lactámicos y tetraciclinas no encontraron relación significativa con la presencia de E. coli resistente a estos antibióticos.
La problemática se intensifica con la presencia de bacterias MDR patógenas fuera del ámbito clínico como en heces de aves de corral, ganado y cerdos, así como directamente en carne y leche crudas y sus derivados por la dificultad que supone el tratamiento de las infecciones que causan y por el impacto epidemiológico debido a la posibilidad de provocar brotes epidémicos como ha sido reportado (21, 27, 39, 40).
Aislamientos MDR de Salmonella se reportó en hisopos cloacales de pollos de engorde de granjas de Malasia (35), y en carnes de pollo y cerdo que se ofrecen en los mercados minoristas de la provincia de Guangdong en China (56).
Es así como Pongsilp et al (68) plantean que debido a que las cepas MDR representan un riesgo de propagación a través de los alimentos deberían ser incluidas en los programas de vigilancia.
Prevalencia de Genes de resistencia a los antibióticos en alimentos de origen animal.
Los genes bla, tet, sul, aac(6')-aph(2") y add fueron los principalmente reportados entre los aislamientos obtenidos de alimentos crudos de origen animal y en las heces de animales de granja que también son reportados en humanos, lo que implica el papel de los alimentos crudos en la diseminación de estos genes de resistencia a través de las cadenas alimentarias o a través del contacto laboral con el animal destinado al consumo humano.
En cuanto a las bacterias-MDR, las publicaciones revisadas describen variados mecanismos de resistencia. Así por ejemplo, en aislamientos de S. aureus obtenidos de hisopados rectales de cerdos en la provincia de Guangdong, China, el fenotipo MDR se explica por la presencia del gen cfr que codifica por una metiltransferasa que modifica la A2503 en el ARNr 23S, confiere resistencia a cinco clases de antibióticos (fenicoles, lincosamidas, oxazolidinonas, pleuromutilinas y estreptogramina A) (39).
La resistencia entre las bacterias-MDR se explica también por la presencia de bombas de eflujo, que son proteínas transportadoras de membrana, organizadas en super familias y tienen la capacidad de expulsar antimicrobianos estructuralmente disímiles. Los aislamientos de S. aureus-MDR en muestras de hisopados rectales de cerdos en porquerizas en Portugal se detectaron los genes mepA, mepR, que codifican para bombas de eflujo de la familia de extrusión de múltiples fármacos y tóxicos (MATE, multidrug and toxic compound extrusion) (25). Entre los aislamientos de Salmonella- MDR que colonizaban a manipuladores de alimentos y de animales de granja (bovinos, pollos y ovejas) en Lagos, Nigeria, el 58,3% de ellos albergaban los genes acrA, acrB y tolC, miembro de la familia de bombas de eflujo de división celular de nodulación de resistencia (RND, resistance-nodulation-cell division)(22).
Los aislamientos resistentes de Campylobacter spp., obtenidos de una instalación de producción intensiva de cerdos en Sudáfrica, se identificó el gen cme B que codifica por la proteína perteneciente a la familia RND (47).
Los elementos genéticos móviles, como plásmidos, transposones e integrones albergan GRAs que facilitan la diseminación de estos genes entre las poblaciones bacterianas y el fortalecimiento de los aislamientos-MDR de origen animal.
Viñes et al (6) reportaron en 18 aislamientos de E. coli resistentes a la colistina (13 de terneros, cuatro de cerdos y uno de un granjero) la detección del gen mcr-1 (confiere resistencia a colistina) asociado con los plásmidos IncX4, IncI2 e IncHI2. Los autores plantean, que la transmisión del gen mcr-1 al agricultor ocurrió por transferencia horizontal desde los aislamientos presentes en los terneros, debido a que los plásmidos eran muy similares (99 % de cobertura, 99,97 % de identidad), lo que destaca el papel del ganado como reservorio de aislamientos con potencial zoonótico (6).
En el caso de los aislamientos de Acinetobacter Baumanii obtenidos de carne cruda de pollo se detectó el gen mcr-1 en un plásmido que se transmite fácilmente entre varias especies Gram negativas, incluidas E. coli, Klebsiella pneumoniae y P. aeruginosa (67). Este hallazgo indica claramente una probable propagación horizontal de determinantes de resistencia a la colistina entre enterobacterias y bacterias no fermentadoras de origen animal.
En aislamientos de E. coli resistentes a quinolonas se detectó GRAs de la familia Qnr (qnrA1, qnrB4 y qnrS1) mediados también por plásmidos, y además, alta prevalencia de estos aislamientos en animales destinados a la producción de alimentos y productos derivados de ellos, lo que potencialmente podría propagar clones de alto riesgo que albergan determinantes de resistencia a quinolonas según lo plantean los autores del estudio (33).
Así mismo, en muestras de queso y carnes crudas de pollo, cerdo y ternera de diversos supermercados de Alemania se aisló E. coli productores de BLEE con el gen bla CTX-M- 1 insertado en un punto caliente para la recombinación del plásmido IncI1, lo que supone que la transmisión del gen podría promoverse aún más (75).
El análisis de secuencia del genoma completo (WGS, por sus siglas en inglés) en plásmidos que portaban GRA en Salmonella spp que se aisló de animales destinados al consumo humano en los Estados Unidos y reveló una alta diversidad genética y el predominio de un tipo de plásmido asociado con los aislamientos de ganado, y otro tipo con aislamientos de pollo (24).
Los integrones se describieron también como transportadores de genes de resistencia, especialmente entre los aislamientos con fenotipo MDR. Así, entre los aislamientos de E. coli resistentes a carbapenemes obtenidos de heces de cerdos en Alemania se detectó el gen blaVIM-1, el cual estaba alojado en un integrón de clase 1 o en el plásmido IncHI2 (65). En los aislamientos de E. coli-MDR circulantes en granjas de cría extensiva de ganado en China se detectó integrones en más del 6% de los casos (30). Sin embargo, los aislamientos de E. coli con características particulares se hacen más estables mostrando baja variabilidad genética. Los aislamientos de E. coli productores de VIM-1 que circularon en granjas avícolas de Alemania se distribuyeron sólo en dos linajes clonales, uno de ellos agrupó los aislamientos que transportaron el gen blaVIM-1 en el plásmido IncHI2 y el segundo linaje agrupó a los aislamientos que portaban el gen en el cromosoma (65).
Estos resultados evidencian la necesidad de emplear medidas estrictas de prevención para evitar la propagación de elementos móviles que transportan determinantes de la resistencia entre los animales destinados al consumo y monitorear su aparición.
Por otro lado, el análisis de la estructura genética de los aislamientos bacterianos permite también establecer el vínculo epidemiológico entre las bacterias de origen zoonótica y humano. Es así como mediante los análisis de tipificación de secuencias multilocus (MLST, por sus siglas en inglés) y electroforesis en gel de campo pulsado (PFGE, por sus siglas en inglés) se determinó los linajes de E. coli que circulaban entre los alimentos minoristas y los humanos en mercados de Egipto. Con el análisis PFGE evidenció una similitud del 75 % entre los aislamientos provenientes de muestras de humanos, carne de res y pollo. Los resultados de MLST estableció seis secuencias tipo (ST) compartidos entre los aislamientos: ST1011, ST156, ST48, ST224 (en pollo y carne de res), ST10 (en humanos y pollo) y ST226 (en humanos y carne de res), evidenciando un posible vínculo epidemiológico con peligros zoonóticos potenciales (43).
Los aislamientos de E. coli-MDR que prevalecieron entre los trabajadores avícolas y las aves de corral en entornos avícolas de Nigeria compartieron los ST48 y ST10 y se observó que dos aislamientos provenientes de fuentes humanas y ambientales que se originaron en la misma granja compartieron un nuevo ST (21).
La estructura genética de la población de K. pneumoniae de cerdos, granjeros, y en individuos que vivían en la misma granja, pero sin contacto con animales reveló una gran diversidad genética con 196 ST distribuidos en cuatro filogrupos (Kp1 a Kp4) y se identificó el intercambio de clones de K. pneumoniae entre cerdos y humanos para los siguientes ST: ST4788, ST661, ST3541 y ST29 (63).
Entre los aislamientos de Salmonella spp. resistentes a los antibióticos se observó baja diversidad genética y alta estabilidad en el tiempo de ciertos clones. La secuenciación del gen fliC de Salmonella spp aislados de granjas de pollos de engorde en Egipto reveló la estrecha identidad de las cepas aisladas con otras cepas de referencia de Salmonella en diferentes países (42). El análisis de las cepas de S. Infantis-MDR recolectadas de pollos de engorde en diferentes años y regiones de Italia, mostró que el 80,5% de los aislados eran BLEE, portadores del gen blaCTX-M1 con un perfil de PFGE prevalente (XbaI.0126). Las cepas restantes tenían una homología genética del 81% al 97% con el perfil XbaI.0126, y se reportaron dos cepas con el perfil XbaI.2621 aisladas de humanos y de heces de aves, confirmando la difusión de este tipo de cepas a lo largo de la cadena alimentaria de los pollos de engorde y en humanos (70).
Así mismo, los aislamientos de Y. enterocolitica en cerdos sacrificados de mataderos de China se caracterizó por patrones de PFGE específicos de la región y confirmó que las cepas aisladas de cerdos están estrechamente relacionadas con las cepas aisladas de infecciones humanas (13).
Esta revisión sistemática tiene las siguientes fortalezas; la búsqueda se limitó a un período que abarcó los últimos siete años (2017-2023), lo que permite al lector comprender la tendencia actual en la resistencia a los antibióticos en bacterias de origen zoonótico. En esta revisión se describen las técnicas más comúnmente empleadas para evaluar la resistencia, las BRAs y GRAs más prevalentes a nivel global y las clases de antibióticos a los que más frecuentemente se reporta resistencia. Además, se revisan los estudios que mediante la tipificación genética demostraron la baja variabilidad genética de los aislamientos zoonóticos, el patrón de diseminación de clones resistentes en diferentes áreas geográficas y la relación genética entre las bacterias zoonóticas con las bacterias de origen humano.
Sin embargo, algunas limitaciones se presentaron. En primer lugar, los artículos incluidos se centraron sobre la literatura científica y no se incluyeron publicaciones gubernamentales que informan sobre este campo. En segundo lugar, no se incluyeron los estudios en acuicultura donde la resistencia ha sido ampliamente reportada. En tercer lugar, en el periodo evaluado no se encontraron publicaciones en Oceanía, lo que limitó el análisis epidemiológico a nivel global. Varios estudios de BRA basaron sus reportes sobre resultados empleando técnicas fenotípicas más que moleculares y no se logró determinar en ellos la estructura genética de las bacterias reportadas y la dinámica de su transmisión.
Esta revisión evidenció alta prevalencia de aislamientos resistentes a los antibióticos, incluidas bacterias MDR. Se destaca E. coli, Salmonella spp. y S. aureus con resistencia a la tetraciclina, la ampicilina/penicilina y el sulfametoxazol/trimetoprima. Estos antibióticos son empleados comúnmente en la medicina humana y veterinaria, lo que limita las opciones de tratamiento, especialmente con la presencia de cepas MDR.
La reducción del consumo de antimicrobianos como la vancomicina en los animales de granja ha sido una de las estrategias que se ha implementado en algunos países, con la reducción significativa de reportes de enterococos resistentes a este antibiótico.
Entre los estudios revisados se encontró alta prevalencia de aislamientos con los GRAs bla, tet y sul frecuentemente transportados por plásmidos o integrones. Estos aislamientos constituyen un reservorio importante de GRAs que pueden diseminarse con facilidad a otras bacterias zoonóticas y a las de origen humano.
Se encontraron estudios que señalaron la baja variabilidad genética entre los aislamientos resistentes de origen zoonótico, con el predominio de uno o dos clones que se mantienen invariables durante décadas. El seguimiento de estos clones en la cadena alimentaria debería ser una prioridad epidemiológica para establecer estrategias de intervención que lleven a reducir los riesgos que representan para la salud pública.
Mejorar la vigilancia epidemiológica de las bacterias zoonóticas resistentes requiere desarrollar pruebas de susceptibilidad antimicrobiana basada en puntos de corte específicos, para su uso en medicina veterinaria e intensificar el empleo de métodos moleculares que permitan conocer la estructura genética y la dinámica de su diseminación.
Los autores declaran no tener ningún conflicto de interés.
Publicado: 07/03/2024
Recibido: 19/10/2023
Aceptado: 23/01/2024